Biologiczne nanostruktury i nanomedycyna
W naszej grupie badawczej stosowane są metody i narzędzia zaawansowanej nanotechnologii do badania struktur biologicznych w nanoskali. Nanostruktury te monitorowane są w żywych komórkach i tkankach, a także w izolowanych układach molekularnych. Uzyskane wyniki służą do opracowywania nowych metod diagnostyki medycznej, farmakologicznego testowania leków, monitorowania toksyczności nanomateriałów, a także do lepszego zrozumienia podstawowych procesów odpowiedzialnych za postęp i leczenie wielu chorób cywilizacyjnych.
Podstawowe cząsteczki życia badane w nanoskali
Cząsteczki o istotnym znaczeniu dla fundamentalnych procesów życiowych: kwasy nukleinowe, białka, lipidy i węglowodany są heterogeniczne w nanoskali. Ta niejednorodność strukturalna determinuje funkcjonalność biomolekuł i wpływa na ich reaktywność. Dlatego struktura chemiczna cząsteczek życia powinna być badana również w nanoskali, najlepiej w warunkach fizjologicznych (Lipiec et al., Angew. Chem. Int. Ed. 2020)
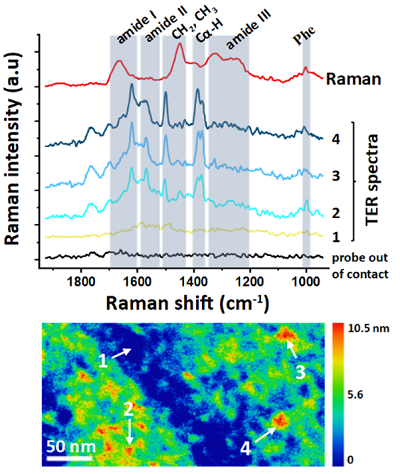
W naszych badaniach stosujemy techniki nanospektroskopii molekularnej: spektroskopię Ramana wzmocnioną na ostrzu sondy skanującej (TERS, ang. tip-enhanced Raman spectroscopy) oraz nanospektroskopię w podczerwieni sprzężoną z mikroskopią skaningową (SPM, ang. scanning probe microscopy) oraz konwencjonalne narzędzia analityczne, w tym spektroskopię Ramana i w zakresie podczerwieni. Nanospektroskopia molekularna łączy nanometryczną rozdzielczość przestrzenną technik SPM oraz selektywność chemiczną spektroskopii cząsteczkowej, dostarczając informacji o strukturze chemicznej i składzie badanych próbek. Schemat układu TERS przedstawiony jest na rys. 1. Modyfikowany próbnik AFM o właściwościach plazmononicznych w kontakcie z powierzchnią próbki prowadzi do wzrostu przekroju czynnego na rozpraszanie Ramana, co skutkuje wzmocnieniem sygnału. Sygnał TERS generowany jest z bardzo małego obszaru, zlokalizowanego w pobliżu ostrza, co umożliwia rejestrowanie widm z nanometryczną rozdzielczością przestrzenną.

Wyniki naszych eksperymentów znacząco poszerzają obecny stan wiedzy i pozwoliły na lepsze zrozumienie procesu agregacji peptydów neurodegeneracyjnych. W szczególności uzyskaliśmy informacje o przestrzennym rozkładzie struktury drugorzędowej białka w nanometrycznych agregatach amyloidu. Przykładowe widma TERS monomerów białka tau (związanych z chorobą Alzheimera) przedstawiono na rys. 2. Zrozumienie wpływu czynników środowiskowych na dystrybucję struktury drugorzędowej warunkującej proces agregacji jest niezbędne do opracowania efektywnej strategii terapeutycznej.
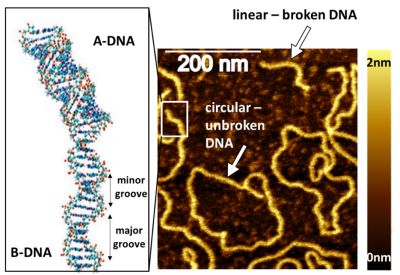
Ponadto, badania prowadzone w naszej grupie skoncentrowane są na monitorowaniu zmian strukturalnych DNA powstałych w wyniku uszkodzeń i towarzyszących im procesów naprawczych. Zrozumienie roli lokalnych zmian konformacyjnych DNA jest fundamentalne m.in. dla pełnego zrozumienia mechanizmów działania leków. Wykazaliśmy, że w miejscu przerwania nici DNA dochodzi do przejścia konformacyjnego z tak zwanego B-DNA (prawoskrętny, 10 par zasad na obrót) do A-DNA (prawoskrętny, 11 par zasad na obrót) (rys. 3). Konformacja DNA warunkuje jego reaktywność w różnych procesach, również naprawczych, gwarantując stabilność materiału genetycznego.
Badanie nanomechaniki komórek i tkanek śródbłonka
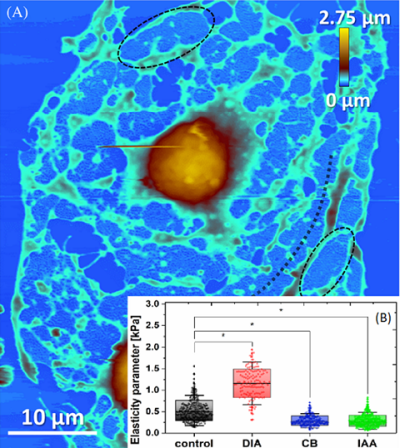
Ze względu na swoje funkcje i lokalizację śródbłonek naczyniowy, czyli zwarta pojedyncza warstwa komórek pokrywająca wewnętrzną stronę naczyń krwionośnych, ulega ciągłemu ściskaniu i rozciąganiu pod wpływem otaczającego środowiska. Znajomość właściwości mechanicznych śródbłonka na poziomie komórkowym jest niezbędna dla dokładnego zrozumienia funkcji i odpowiedzi mechanicznej komórek zarówno na środowisko naturalne, jak i czynniki patologiczne. Prowadzimy badania eksperymentalne nad nanomechaniczną odpowiedzią komórek śródbłonka na zapalenie wywołane czynnikami środowiskowymi i/lub wybranymi związkami chemicznymi. W naszych badaniach wykorzystujemy spektroskopię sił w celu mapowania właściwości mechanicznych badanych próbek. Analizujemy zmiany właściwości mechanicznych komórek śródbłonka spowodowane stanem zapalnym wywołanym chorobami cywilizacyjnymi, takimi jak hiperglikemia, miażdżyca tętnic czy niealkoholowe stłuszczenie wątroby (rys. 4).
Obrazowanie porowatych nanostruktur biologicznych
Nasza grupa koncentruje się również na badaniach komórek śródbłonka wątroby (ang. the liver sinusoidal endothelium LSEC). Cechą charakterystyczną dla komórek LSEC jest obecność fenestracji - otwartych porów, w których tworzeniu uczestniczy cytoszkielet komórki. Liczba i średnica porów determinują dwukierunkowy transport między krwią a hepatocytami. Typowa średnica fenestracji mieści się w przedziale od 50 do 300 nanometrów, jej precyzyjne wyznaczenie jest dużym wyzwaniem eksperymentalnym. Nasze badania obejmują pionierskie zastosowanie mikroskopii sił atomowych w celu mapowania własności mechanicznych żywych komórek i wizualizacji fenestracji (zob. Zapotoczny i in., Hepatology, 2019; DOI:10.1002/hep.30232). Śledzenie dynamiki zmian rozmiarów fenestracji w żywych komórkach LSEC za pomocą AFM pozwala na badanie zależnych od czasu zmian fenotypu związanych z chorobami i/lub różnymi niekorzystnymi czynnikami środowiskowymi, a także spowodowanych farmakoterapią.
Nanomechanika komórki – analiza danych eksperymentalnych
Badamy właściwości fizyczne układów biologicznych, takie jak elastyczność. Elastyczność jest ściśle związana z funkcjonalnymi lub patologicznymi zmianami w komórkach. Koncentrujemy się na właściwej interpretacji otrzymywanych danych eksperymentalnych. Dwa najczęściej stosowane podejścia obejmują jednoparametrowe modele Hertza i Sneddona. W naszym podejściu interpretujemy eksperymentalną krzywą siła-odległość w bardziej złożony sposób, aby określić parametr sprężystości, który zmienia się wraz z głębokością indentacji. Najlepsze przybliżenie tej właściwości znajdujemy, stosując model dwuwarstwowy z czterema efektywnymi parametrami, co daje nam lepsze zrozumienie złożonej struktury komórek i ich właściwości fizycznych.
Realizacja projektu badawczego
Finansowanie:

Podczas realizacji zadań badawczych zaplanowanych w ramach projektu OPUS 16, nasza grupa badawcza przeprowadziła kompleksowe badania nad przejściami strukturalnymi w DNA na różnych poziomach organizacji (cząsteczki, chromosomy oraz jądra komórkowe). W szczególności, zbadaliśmy przerwania nici DNA spowodowane działaniem leku przeciwnowotworowego (bleomycyna) stosując nowoczesne techniki mikroskopowe oraz spektroskopowe. Wyniki przeprowadzonych badań przedstawiono poniżej.
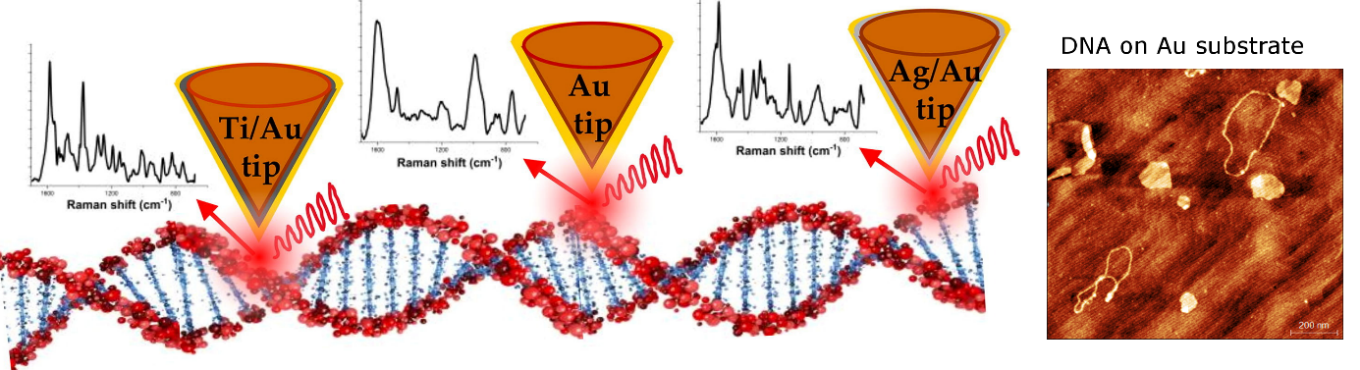
Optymalizacja preparatyki próbników TERS dedykowanych do badań struktury cząsteczek DNA
Nasza grupa badawcza zoptymalizowała procedurę otrzymywania modyfikowanych próbników stosowanych w badaniach cząsteczek DNA za pomocą techniki TERS. W tym celu, przetestowaliśmy różnorodne metody umożliwiające przygotowanie zarówno modyfikowanych próbników, jak i podłoży służących do obrazowania i chemicznej identyfikacji delikatnych struktur biologicznych, takich jak DNA. Przykładowe wyniki zamieszczono na Rys. 6. Przeprowadzone badania stanowią doskonały punkt wyjścia dla dalszych prac skupionych na śledzeniu dynamiki zmian konformacji DNA podczas oddziaływania z innymi cząsteczkami, np. białkami naprawczymi. Więcej szczegółowych informacji znajduje się w publikacji: Spectrochim. Acta A Mol. Biomol. Spectrosc. 281, 121595
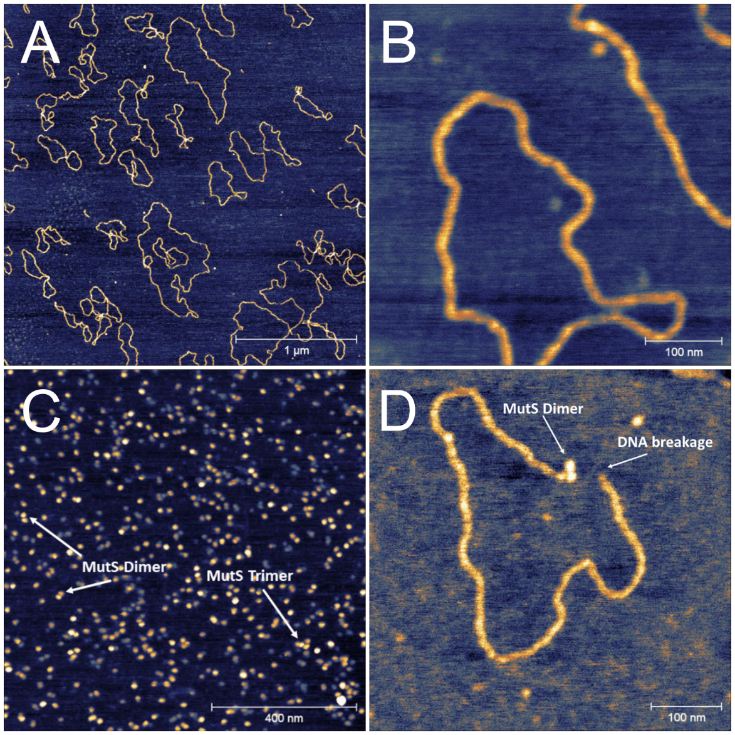
Oddziaływanie DNA z białkami naprawczymi
Zbadaliśmy dynamikę zmian konformacyjnych DNA podczas procesu naprawy mutacji punktowych (niekomplementarnych par zasad) z zastosowaniem mikroskopii sił atomowych oraz metod spektroskopowych. Zastosowanie techniki AFM umożliwiło zobrazowanie struktury DNA oraz białka naprawczego MutS w symulowanych warunkach fizjologicznych. Istotną rolę w ww. badaniach odegrało zastosowanie PeakForce Tapping – nowoczesnego trybu obrazowania umożliwiającego zwizualizowanie struktury DNA przy użyciu minimalnych sił oddziaływania próbka – ostrze próbnika. Przykładowe obrazy uzyskane w ten sposób przedstawiono na Rys. 7. Właściwości strukturalne DNA i białka MutS otrzymane w wyniku analizy obrazów umożliwiłu poprawną interpretację danych spektroskopowych uzyskanych w równoległych eksperymentach (spektroskopia Ramana oraz spektroskopia w podczerwieni). Technika AFM umożliwiła również zwizualizowanie procesu tworzenia kompleksu DNA – MutS. Wykazaliśmy, że dimeryzajca białka MutS jest niezbędna do zainicjowania procesu naprawy, a stężenie dimerów MutS w układzie jest ściśle zależne od stężenia jonów Mg2+. Udowodniliśmy, że w dalszym etapie procesu naprawczego dochodzi do przerwania podwójnej nici DNA, co jest niezbędne do usunięcia mutacji punktowej.
Zmiany molekularne i morfologiczne w izolowanych chromosomach
Kolejnym obiektem naszych badań były chromosomy izolowane z komórek nowotworowych (HeLa). Chromosomy to struktury biologiczne, o charakterystycznym kształcie litery X, składające się z wysoce upakowanego materiału genetycznego oraz białek stabilizujących. W ramach zaplanowanych zadań, nasza grupa zbadała zmiany w strukturze chemicznej oraz morfologii chromosomów inkubowanych z bleomycyną – lekiem przeciwnowotworowym znanym z indukowania pojedynczych oraz podwójnych przerwań nici DNA. W przypadku gdy ww. przerwania nie zostaną naprawione wystarczająco szybko, nastąpują nieodwracalne zmiany w strukturze chromosomów – tzw. aberracje chromosomowe. W celu zbadania wpływu bleomycyny na występowanie aberracji zastosowaliśmy dwie komplementarne techniki: mikroskopię sił atomowych (AFM) oraz mikrospektroskopię Ramana. AFM umożliwiła zobrazowanie zmian w morfologii chromosomów (Rys. 8), natomiast mikrospektroskopia Ramana pozwoliła na określenie zmian chemicznych zachodzących w strukturze chromatyny.

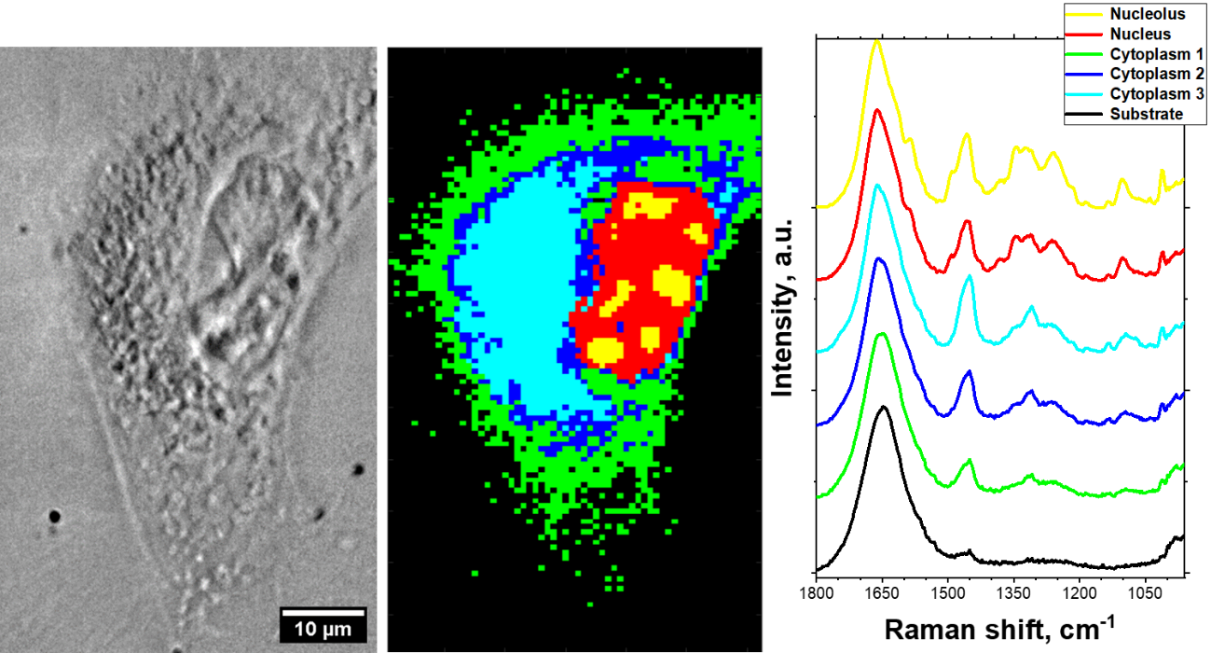
Odpowiedź biochemiczna żywych komórek na zmiany wywołane uszkodzeniem DNA
Stosując mikrospektroskopię Ramana zbadaliśmy wpływ przerwań nici DNA na zmiany w biochemii żywych komórek. Otrzymane wyniki poddaliśmy wielowymiarowej analizie statystycznej metodami hierarchicznej analizy klastrowej (ang. Hierarchical Clustering Algorithm, HCA) oraz analizy głównych składowych (ang. Principal Component Analysis, PCA). Zastosowanie metod statystycznych pozwoliło na wyodrębnienie obszarów komórkowych różniących się charakterystyką spektralną, a więc także i składem chemicznym, co stanowi swoisty biochemiczny odcisk palca wywołanych zmian. Wyodrębnione obszary odpowiadają różnym organellom komórkowym, w tym istotnym dla naszych badań jądrom komórkowym (Rys. 9). Zastosowanie takiego podejścia umożliwiło monitorowanie systematycznych zmian spektralnych związanych z odpowiedzią biochemiczną na działanie bleomycyny w różnych stężeniach. Szczegółowe wyniki przeprowadzonych badań przedstawiono w publikacji: Int. J. Mol. Sci. 2022, 23(7), 3524.
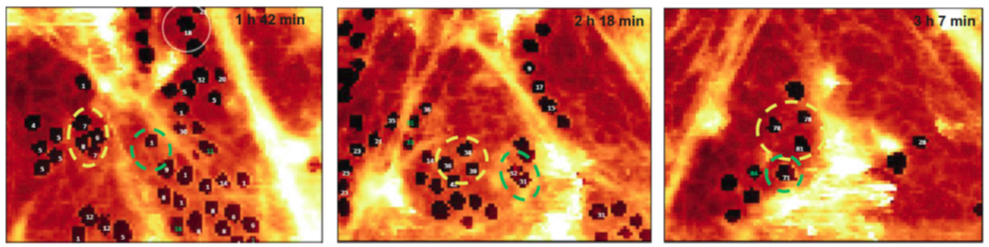
Obrazowanie procesów naprawczych w chromatynie za pomocą mikroskopii fluorescencyjnej
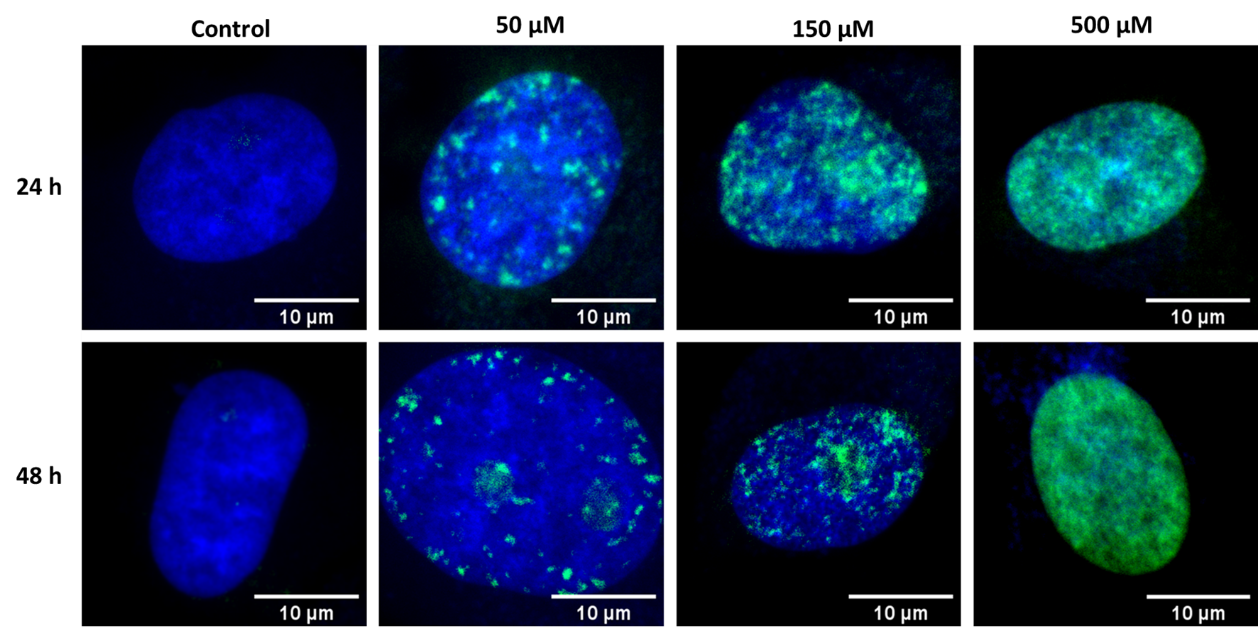
Zastosowaliśmy obrazowanie fluorescencyjne by zaobserwować naprawę uszkodzonej chromatyny. Wizualizacja procesu naprawy była możliwa dzięki zastosowaniu barwników fluorescencyjnych wiążących się selektywnie do jądra komórkowego (DAPI) lub fosforylowanego histonu H2AX (Alexa Fluor 488). Fosforylacja histonu H2AX jest typową oznaką zainicjowania procesu naprawy przerwań nici DNA, w związku z tym, zastosowanie znakowanego fluorescencyjnie przeciwciała na H2AX umożliwia zlokalizowanie i zliczenie uszkodzeń DNA na podstawie analizy ognisk. Jak zaobserwowaliśmy, liczba uszkodzeń chromatyny rosła systematycznie wraz ze wzrostem stężenia stosowanego chemoterapeutyku. Szczegóły przeprowadzonych badań znajdują się w: Int. J. Mol. Sci. 2022, 23(7), 3524
.
- dr Karol Cieślik
- mgr Michał Czaja
- dr Ewelina Lipiec
- dr Kamila Sofińska
- lic. Marta Urbańska
- dr Natalia Wilkosz
- prof. dr hab. Marek Szymoński
Członkowie grupy
- dr hab. Ewelina Lipiec - kierownik
- prof. dr hab. Marek Szymoński
- dr. Dawid Lupa
- dr. Kamila Sofińska
- dr. Natalia Wilkosz
- mgr Michał Czaja
- mgr Sara Seweryn
- mgr Katarzyna Skirlińska
- lic. Marta Urbańska
 dr hab. Ewelina Lipiec
dr hab. Ewelina Lipiec